RFantibody로 de novo antibody discovery
RFantibody는 구조 기반으로 새로운 항체와 나노바디를 디자인 할 수 있는 소프트웨어입니다. 구체적으로 RFantibody는 아래 세 가지 독립적인 소프트웨어들로 구성되어 있습니다.
- RFdiffusion의 단백질 알파 카본에 대한 골격을 디자인 합니다.
- ProteinMPNN을 사용해 위에서 디자인한 골격에 그럴듯한 아미노산 서열을 생성합니다.
- RoseTTAFold2으로 생성된 서열의 단백질 구조를 만들어보고 잘못된 디자인은 필터링합니다.
이런 RFantibody 파이프라인에 대한 자세한 내용은 공개 논문에서 확인할 수 있습니다. 또한 RFantibody는 MIT 라이선스로 비영리 및 영리 사용자 모두에게 무료입니다.
1 설치 및 사전 준비
1.1 Docker와 GPU
RFantibody는 Docker 컨테이너에서 실행되도록 설계되었습니다. Docker 컨테이너는 호스트 운영 체제 위에 별도의 운영 체제를 실행해서 다음과 같은 장점을 제공합니다.
- 설치 간소화: Docker 소프트웨어 제품군만 있으면 됩니다.
- 호스트 시스템 독립성: 컨테이너 내부에서 실행되므로 어디에서 실행하든 기본적으로 동일하게 작동합니다.
Docker를 설치하는 방법은 공식 홈페이지를 참고하세요. 이미 Docker가 설치되어 있는지 여부는 터미널에서 which docker 로 확인할 수 있습니다. 이 명령이 경로를 반환하면 Docker를 사용할 수 있으며 실행할 준비가 된 것입니다.
또한 RFantibody를 실행하려면 NVIDIA GPU가 필요합니다. 다음을 실행하여 NVIDIA GPU를 사용할 수 있는지 확인할 수 있습니다. nvidia-smi 이 명령이 성공적으로 실행되면 호환되는 GPU가 있는 것이며 RFantibody를 실행할 수 있습니다.
1.2 가중치 다운로드
먼저 Git을 사용하여 RFantibody 저장소 클론하고 다운로드된 디렉토리로 이동합니다. 그런 모델의 가중치를 RFantibody/weights 디렉토리에 다운로드합니다.
git clone https://github.com/partrita/RFantibody
cd RFantibody/
bash include/download_weights.sh1.3 RFantibody Docker 컨테이너 빌드 및 실행
Docker 컨테이너를 시작할 올바른 권한이 있는지 확인하려면 다음을 실행해야 합니다.
sudo usermod -aG docker $USER이 명령을 실행한 후 변경 사항이 적용되도록 터미널 세션을 다시 시작해야 합니다.
1.4 Docker 이미지 빌드 및 시작하기
RFantibody가 다운로드된 디렉토리에서 다음 명령을 실행하여 RFantibody용 Docker 이미지를 빌드합니다.
docker build -t rfantibody .방금 빌드한 이미지를 기반으로 Docker 컨테이너를 시작하려면 다음 명령을 실행합니다.
docker run --name rfantibody --gpus all -v .:/home -it rfantibody이렇게 하면 현재 폴더를 Docker 컨테이너의 /home 폴더와 연결 할 수 있습니다.
1.5 Python 환경 설정
RFantibody 컨테이너에서 다음 스크립트를 실행해 Python 의존성 라이브러리를 설치합니다. 구체적으로 다음이 설치 됩니다.
- Python 환경 빌드 준비를 위해 Deep Graph Library를 다운로드합니다.
- Poetry를 사용하여 Python 환경을 빌드합니다.
- USalign 실행 파일을 빌드합니다.
bash /home/include/setup.sh1.6 HLT 파일 형식
RFantibody는 각 단계 간에 PDB 파일 구조를 전달해야 합니다. 그리고 PDB 파일 구조에는 다음이 포함되어 있어야 합니다.
- 현재 디자인 중인 항체-표적 복합체 구조 정보
- 어떤 사슬이 중쇄(Heavy chain), 경쇄(Light chain) 및 표적 사슬(Target chain)인지 여부
- 어떤 잔기가 어떤 CDR 루프에 있는지 여부
파이프라인 단계 간에 이 정보를 전달할 수 있도록 HLT 파일이라고 하는 새로운 파일 형식을 정의해야 합니다. HLT 파일은 기본적으로 .pdb 파일이지만 다음과 같은 수정사항이 있습니다.
- 중쇄는 사슬 ID ’H’로 표시됩니다.
- 경쇄는 사슬 ID ’L’로 표시됩니다.
- 표적 사슬은 (표적 사슬이 여러 개라도) 사슬 ID ’T’로 표시됩니다.
- 파일의 사슬 순서는 중쇄, 경쇄, 표적 순입니다.
- 파일 끝에는 각 CDR 루프 기반 절대(사슬별이 아닌) 아미노산 인덱스를 나타내는 PDB Remark가 있습니다.
1.6.1 HLT 파일로 변환하기
RFantibody는 입력으로 HLT 파일 변환을 수행하는 스크립트를 /scripts/util/chothia2HLT.py에 제공합니다.
# rfantibody 컨테이너 내부에서
poetry run python /home/scripts/util/chothia2HLT.py \
-i /home/scripts/examples/rfdiffusion/example_inputs/8tlm_chothia.pdb \
-o /home/scripts/examples/rfdiffusion/example_inputs/8tlm \
-H A -L B -T C이 스크립트는 Chothia 주석이 달린 .pdb 파일을 예상합니다. 이러한 파일의 훌륭한 소스는 PDB의 모든 항체 및 나노항체의 Chothia 주석 구조를 제공하고 몇 달마다 업데이트되는 SabDab입니다.
또한 RFantibody 공개 논문에서 사용된 HLT 형식의 항체 및 나노바디 프레임워크는 다음 위치에 포함되어 있습니다.
- 나노바디 프레임워크:
/scripts/examples/example_inputs/h-NbBCII10.pdb - 항체 프레임워크:
/scripts/examples/example_inputs/hu-4D5-8_Fv.pdb
2 사용법
앞서 이야기 했듯이 RFantibody는 3단계를 거쳐 작동합니다. 각 단계마다 명령어를 입력하는 방법은 이후에 설명하도록 하고 여기서는 사용자의 편의를 위해 작성한 bash 스크립트를 먼저 살펴보겠습니다.
2.1 한번에 실행 하기
실행 방법은 아래와 같습니다.
# rfantibody 컨테이너 내부에서
bash /home/scripts/examples/rfdiffusion/example_9gox.sh텍스트 에디터로 해당 스크립트를 열어보면 아래와 같습니다. 상단의 스크립트의 변수를 원하는 값으로 수정해서 사용하도록 하세요.
#!/bin/bash
# Define common variables
NAME="9gox"
PDB_ANTIBODY="CCR8_subset.pdb"
PDB_ANTIGEN="9gox_CLC.pdb"
HOTSPOT="A175, S176, G179, V180, L181"
DESIGN_LOOPS="H1:7,H2:5,H3:5-8"
NUM_DESIGNS=2
SEQS_PER_STRUCT=2
# Define directory structure
BASE_DIR="/home/scripts"
INPUT_DIR="${BASE_DIR}/examples"
OUTPUT_DIR="${INPUT_DIR}"
RF_DIFFUSION_OUTPUT="${OUTPUT_DIR}/rfdiffusion/example_outputs/${NAME}"
PROTEINMPNN_OUTPUT="${OUTPUT_DIR}/proteinmpnn/example_outputs/${NAME}"
RF2_OUTPUT="${OUTPUT_DIR}/rf2/example_outputs/${NAME}"
# [이후 코드 생략]2.2 Fine-tuned RFdiffusion
RFantibody의 첫 번째 단계인 항체 맞춤형 RFdiffusion 버전을 사용하는 방법을 살펴보겠습니다. 이 단계는 항체와 표적 단백질의 도킹 구조 파일을 생성합니다. 다음은 RFdiffusion을 실행하는 예제 명령입니다.
# rfantibody 컨테이너 내부에서
poetry run python /home/rfantibody/scripts/rfdiffusion_inference.py \
--config-path src/rfantibody/rfdiffusion/config/inference \
--config-name antibody \
antibody.target_pdb=/home/scripts/examples/example_inputs/rsv_site3.pdb \
antibody.framework_pdb=/home/scripts/examples/example_inputs/hu-4D5-8_Fv.pdb \
inference.ckpt_override_path=/home/weights/RFdiffusion_Ab.pt \
'ppi.hotspot_res=[T305,T456]' \
'antibody.design_loops=[L1:8-13,L2:7,L3:9-11,H1:7,H2:6,H3:5-13]' \
inference.num_designs=20 \
inference.output_prefix=/home/scripts/examples/example_outputs/ab_des위 명령어에 대해 좀 더 자세히 설명해보겠습니다.
antibody.target_pdb: 항체를 디자인하려는 표적 구조의 경로입니다. 일반적으로 파이프라인 실행의 계산 비용을 줄이기 위해 잘린 표적 구조입니다. 절단 전략은 여기에서 자세히 설명합니다.antibody.framework_pdb: 디자인에 사용하려는 HLT 형식의 항체 프레임워크 경로입니다. RFdiffusion은 루프로 주석이 달린 프레임워크 영역의 구조와 서열만 디자인합니다. 이렇게 하면 이미 최적화된 프레임워크의 도킹 및 루프를 디자인할 수 있습니다.inference.ckpt_override_path: 추론에 사용할 RFdiffusion 모델 가중치 세트의 경로입니다.ppi.hotspot_res: 에피토프를 정의하는 핫스팟 잔기 목록입니다. 일반 RFdiffusion과 동일한 형식으로 제공됩니다. 핫스팟 선택에 대한 자세한 내용은 여기에서 설명합니다.antibody.design_loops: 각 CDR 루프를 허용된 루프 길이 범위에 매핑하는 사전입니다. 각 루프의 길이는 이 범위에서 균일하게 샘플링되며 다른 루프에 대해 샘플링된 길이와 독립적으로 샘플링됩니다. CDR 루프가 프레임워크에 있지만 사전에 없는 경우 이 CDR 루프는 디자인 중에 서열 및 구조가 고정됩니다. CDR 루프가 사전에 포함되어 있지만 길이 범위가 제공되지 않은 경우 이 CDR 루프는 서열 및 구조가 디자인되지만 프레임워크 구조에 제공된 루프 길이로만 디자인됩니다.inference.num_designs: 생성해야 하는 디자인 수입니다.inference.output_prefix: 생성할.pdb파일 출력의 접두사입니다.
2.3 ProteinMPNN
두 번째 단계는 윗 단계 RFdiffusion에서 생성된 구조 파일을 가져와 CDR 루프에 서열을 할당하는 것입니다. ProteinMPNN은 다음 명령을 사용하여 HLT 형식의 .pdb 파일 디렉토리에서 실행될 수 있습니다.
# rfantibody 컨테이너 내부에서
poetry run python /home/scripts/proteinmpnn_interface_design.py \
-pdbdir /home/scripts/examples/example_outputs/ab \
-outpdbdir /home/scripts/examples/example_outputs/ab2.4 RoseTTAFold2
마지막 단계는 항체 맞춤형 RoseTTAFold2를 사용해 방금 디자인한 서열의 구조를 예측하는 것입니다. 그런 다음 해당 항체와 타겟 단백질이 결합될 것이라고 확신하는지 평가합니다. 간단하게 다음 명령을 통해 실행할 수 있습니다.
# rfantibody 컨테이너 내부에서
poetry run python /home/scripts/rf2_predict.py \
input.pdb_dir=/path/to/inputdir \
output.pdb_dir=/path/to/outputdir3 분석 결과
앞서 이야기 했듯이 RFantibody는 3단계에 이뤄 실행되며 각각의 결과에 대해 살펴보겠습니다.
3.1 Fine-tuned RFdiffusion
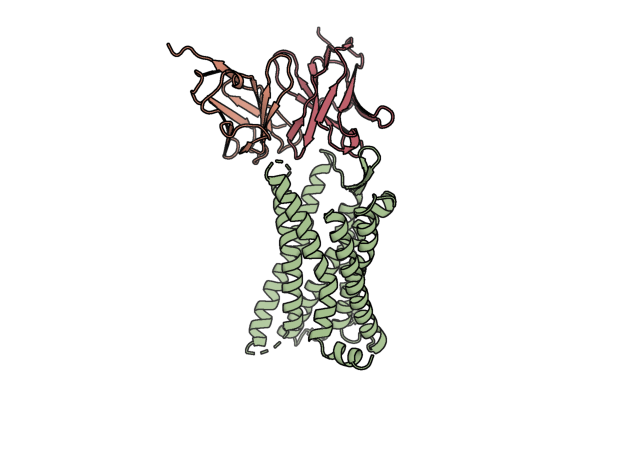
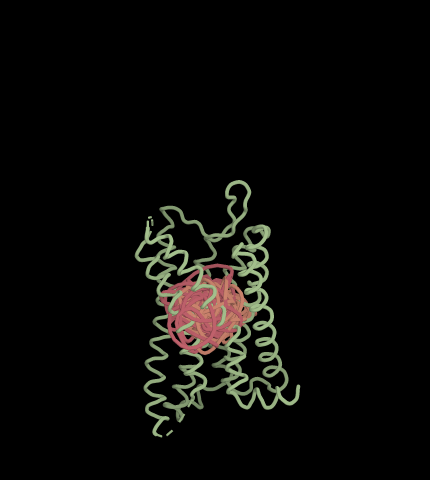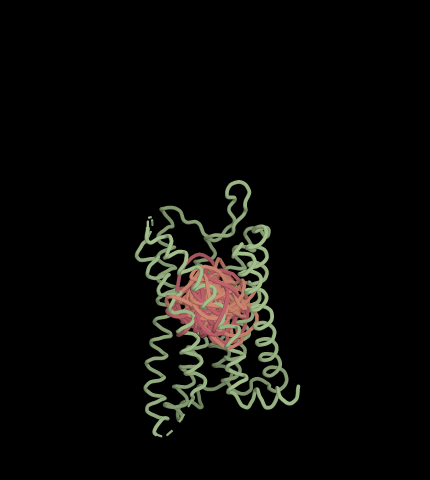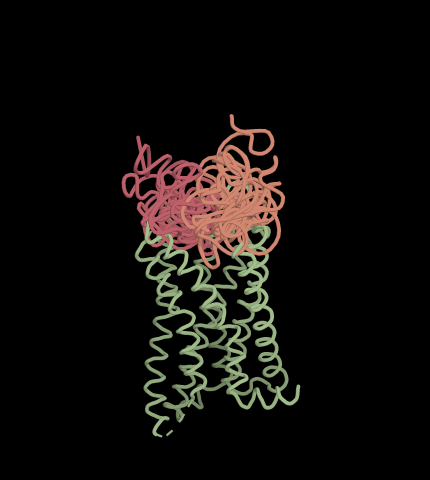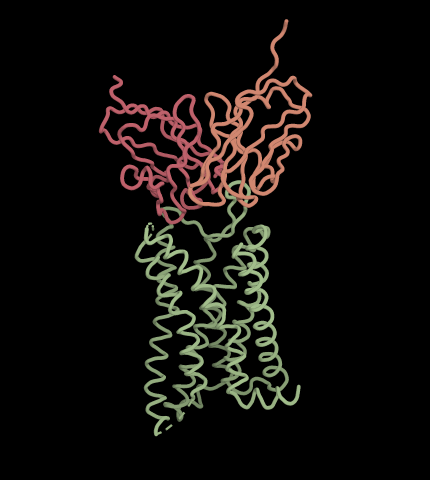
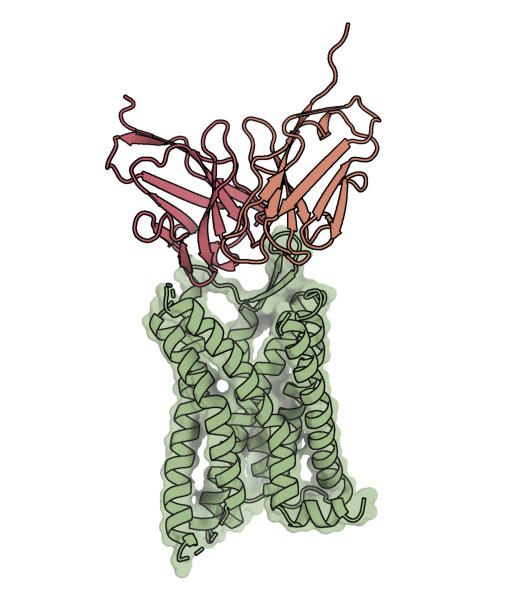
첫 번째 단계인 RFdiffusion를 통해 아래 그림과 같이 타겟 단백질에 결합하는 항체 VH, VL 구조를 형성 할 수 있습니다.
다만 생성된 항체 구조에는 아미노산 residue에 대한 정보는 없고 Carbon alpha에 대한 위치 정보만 들어 있습니다. 따라서 다음 단계인 ProteinMPNN을 통해 구조에 맞는 아미노산 서열을 생성해야 합니다.
3.2 ProteinMPNN
ProteinMPNN을 실행하면 아래 예시와 같은 서열 정보가 포함된 .pdb파일이 만들어집니다. 이렇게 만들어진 서열이 진짜 앞에서 생성한 구조 파일에 적합한지 평가를 위해 다음 단계인 Protein folding이 필요합니다.
>ab_0_dldesign_0_H
QVQLVQSGAEVKKPGSSVKVSCKAVGASISSGIINWVRQAPGQGLEWMGRVDRSGGAYYAQKFQGRVTIT
ADESTSTAYMELRSEDTAVYYCALEVNGYLTGWGQGTLVTVSSAS
>ab_0_dldesign_1_H
QVQLVQSGAEVKKPGSSVKVSCKAVGDSISSGIINWVRQAPGQGLEWMGRVDRAGGAYYAQKFQGRVTIT
ADESTSTAYMELRSEDTAVYYCALEVNGYLTGWGQGTLVTVSSAS
>ab_0_dldesign_2_H
QVQLVQSGAEVKKPGSSVKVSCKAVGDSISSGIINWVRQAPGQGLEWMGRVDRSGGAYYAQKFQGRVTIT
ADESTSTAYMELRSEDTAVYYCALEVNGYLTGWGQGTLVTVSSAS
>ab_0_dldesign_3_H
QVQLVQSGAEVKKPGSSVKVSCKAVGASISSGIINWVRQAPGQGLEWMGRVDRSGGAYYAQKFQGRVTIT
ADESTSTAYMELRSEDTAVYYCALEVNGYLTGWGQGTLVTVSSAS
>ab_0_dldesign_4_H
QVQLVQSGAEVKKPGSSVKVSCKAVGASISSGIINWVRQAPGQGLEWMGRVDRSGGAYYAQKFQGRVTIT
ADESTSTAYMELRSEDTAVYYCALEVNGYLTGWGQGTLVTVSSAS
>ab_0_dldesign_5_H
QVQLVQSGAEVKKPGSSVKVSCKAVGASISSGIINWVRQAPGQGLEWMGRVDRAGGAYYAQKFQGRVTIT
ADESTSTAYMELRSEDTAVYYCALEVNGYLTGWGQGTLVTVSSAS3.3 RoseTTAFold2
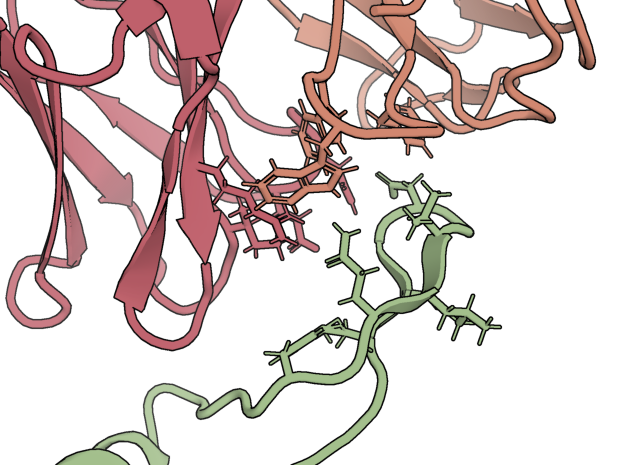
RoseTTAFold2의 결과를 확인해보면 아래와 같이 아미노산의 서열이 들어가 있는 것을 확인 할 수 있습니다.
이렇게 항체와 타겟 단백질의 결합 구조 파일을 생성하는 것으로 De novo antibody discovery가 완료되었습니다.
4 마치며
de novo 항체 설계는 아직 초기 기술입니다. 그래서 실제로 결합하는 항체를 찾는 과정은 어려운 일입니다. 그러나 앞으로 더 많은 항체 설계 작업이 수행되고 모범 사례가 쌓이면 이 기술이 완벽해질 것입니다. 현재까지 알려진 de novo 항체 설계를 위한 고려 사항은 아래와 같습니다.
4.1 표적 부위 선택
단백질의 모든 부위가 항체에 결합할 수 있는 것은 아닙니다. 즉, 결합에 적합한 후보가 되려면 해당 부위에 결합제가 상호작용할 수 있는 최소 3개 이상의 소수성 잔기가 있어야 합니다. 그리고 전하를 띤 극성 아미노산에 대한 결합은 여전히 상당히 어렵습니다. 또한 당화가 일어나는 부위에 대한 결합도 어렵습니다. 그리고 구조가 유연한 루프에 대한 결합도 역사적으로 어려운 부위이지만 RFdiffusion을 사용한 논문도 존재합니다.
4.2 나노바디 도킹 문제
de novo 나노바디 설계를 해보면 많은 경우 CDR이 아닌 측면으로 도킹하는 것을 볼 수 있습니다. 이는 오류가 아니며 실제로 나노바디가 종종 이런 측면으로 결합하기 때문입니다. 따라서 핫스팟과 CDR 길이를 조정한다면 항체와 비슷한 도킹 결과를 얻을 수 있습니다. 다만 항체와 유사한 도킹을 원한다면 항체 프레임워크를 사용하여 설계하는 편이 더 좋은 접근법입니다.
4.3 표적 단백질 잘라내기
RFdiffusion과 RF2의 계산 시간은 시스템의 잔기 수 N에 대해 \(O(N^2)\)로 증가합니다. RFantibody 파이프라인의 모든 단계는 잘라낸 표적 단백질을 허용하도록 설계되었고 따라서 표적 단백질의 크기가 큰 경우 계산량을 줄이기 위해 불필요한 서열은 잘라내는 것이 좋습니다. 물론 표적 단백질의 어느 부분을 잘라낼 것인가는 사용자에 따라 의견이 다를 것입니다. 일반적으로는 2차 구조를 형성하는 부위는 피하고 링커로 연결된 지점을 잘라내는 것이 추천됩니다. 그러나 바이러스 스파이크 단백질과 같은 단백질은 이런 점이 덜 명확할 수 있습니다. 그러니 PyMol을 사용해 직접 눈으로 보고 표적 단백질을 잘라내는 것을 추천합니다. 또한 원하는 표적 부위(핫스팟) 주변 10Å의 단백질은 남겨두는 것을 권장합니다.
4.4 결합 위치(핫스팟) 고르기
결합 위치(핫스팟)은 항체가 표적 단백질에 상호작용하는 부분입니다. RFantibody 모델의 학습 과정에서 핫스팟은 항체의 CDR 잔기와 Cβ 거리가 8Å 미만인 부위로 지정되었습니다. RFantibody는 일반 RFdiffusion보다 선택된 핫스팟에 더 민감한 것으로 나타났고 부적절한 핫스팟이 지정된 경우 도킹되지 않은 항체를 생성합니다. 따라서 수천 개의 항체를 생성하기 전에 소수의 항체 생성을 실험해보고 지정한 핫스팟이 적절한지 판단해보세요.
4.5 항체 설계 규모에 대하여
논문에서 보고된 일부 표적 단백질의 경우 단지 95개만 설계했었어도 VHH 결합체를 식별할 수 있었습니다. 그러나 일반적으로는 1만개 정도의 항체 서열을 생성해야 할 것으로 예상합니다. 왜냐하면 현재 신뢰할 수 있는 in silico 필터링 지표가 없기 때문입니다. 현재는 긍정적이든 부정적이든 항체 설계를 통해 얻은 데이터가 부족하고 앞으로 더 많은 실험 데이터가 커뮤니티에 공유 되어야 합니다. 그러면 더 신뢰할 수 있는 in silico 필터를 만들 수 있을 것입니다. 그러면 더 높은 성공률을 통해 저렴한 항체 설계를 할 수 있을 것입니다.
4.6 CDR 길이 선택
우리의 설계 캠페인에 사용된 루프 범위는 RFdiffusion 예제 파일에 제공되어 있습니다. 이 범위는 각 루프의 자연 발생 길이 빈도를 살펴보고 대부분의 밀도를 포함하도록 결정했습니다. 또한 비교적 짧은 H3 루프를 선택하려고 했는데, 이는 설계와 예측이 더 쉬우면서도 효과적으로 결합할 수 있는 충분한 길이를 제공할 것이라고 판단했기 때문입니다. 단백질의 소수성 포켓을 표적으로 할 때는 길이가 긴 H3 서열이 유용할 것입니다. 따라서 이런 경우에는 예제에서 제공하는 것보다 H3 범위를 늘려야 합니다.
4.7 in silico 필터링 전략
다음과 같은 최소한의 필터링 기준을 권장합니다
- RF2 pAE < 10
- RMSD(디자인과 RF2 예측결과의 차이) < 2Å
- Rosetta ddG < -20
현재 RFantibody 파이프라인의 가장 큰 문제는 효과적인 in silico 필터가 없다는 것입니다. 그러니 더 많은 실험을 통해 데이터를 수집할 필요가 있습니다.